Beitrag in Eurosurveillance veröffentlicht (27.10.2022)
Im Rahmen von GenSurv und MolTrax konnte die erfolgreiche Anwendung des integrierten genomischen Surveillance in Düsseldorf zur Verfolgung von SARS-CoV-2-Übertragungsketten in der Bevölkerung sowie zur Erkennung und Untersuchung von reiseassoziierten SARS-CoV-2-Infektionsclustern demonstriert werden. Ein Artikel dazu wurde Ende im Oktober 2022 in dem Fachjournal Eurosurveillance veröffentlicht.
„Wo stecken sich die Menschen eigentlich an?“ Dieser Frage versuchen sich im Netzwerk Universitätsmedizin (NUM) aktive Düsseldorfer Forscher*innen seit Beginn der Corona-Pandemie mit modernen Methoden der genomischen Epidemiologie anzunähern.
In einer aktuellen Publikation [1] zeigen die Wissenschaftler*innen nun, dass das von ihnen entwickelte Düsseldorfer System der „integrierten genomischen Surveillance“ [2] tatsächlich das Zurückverfolgen von Infektionsketten in der Bevölkerung ermöglicht – und damit Rückschlüsse erlaubt z. B. über die Rolle von Reiserückkehrern im Infektionsgeschehen.
Zuverlässige Daten über die Ausbreitung von Krankheitserregern in der Bevölkerung sind nicht nur von wissenschaftlichem Interesse, sondern auch für die Infektionsprävention – also das Verhindern von Infektionen, z. B. durch gezielte Gegenmaßnahmen – hochgradig relevant.
Im Düsseldorfer System (siehe Abbildung 1) werden dabei moderne genomische Methoden kombiniert mit klassischer Epidemiologie und Kontaktnachverfolgungsdaten des Düsseldorfer Gesundheitsamtes, um Infektionsketten besser nachvollziehen zu können.
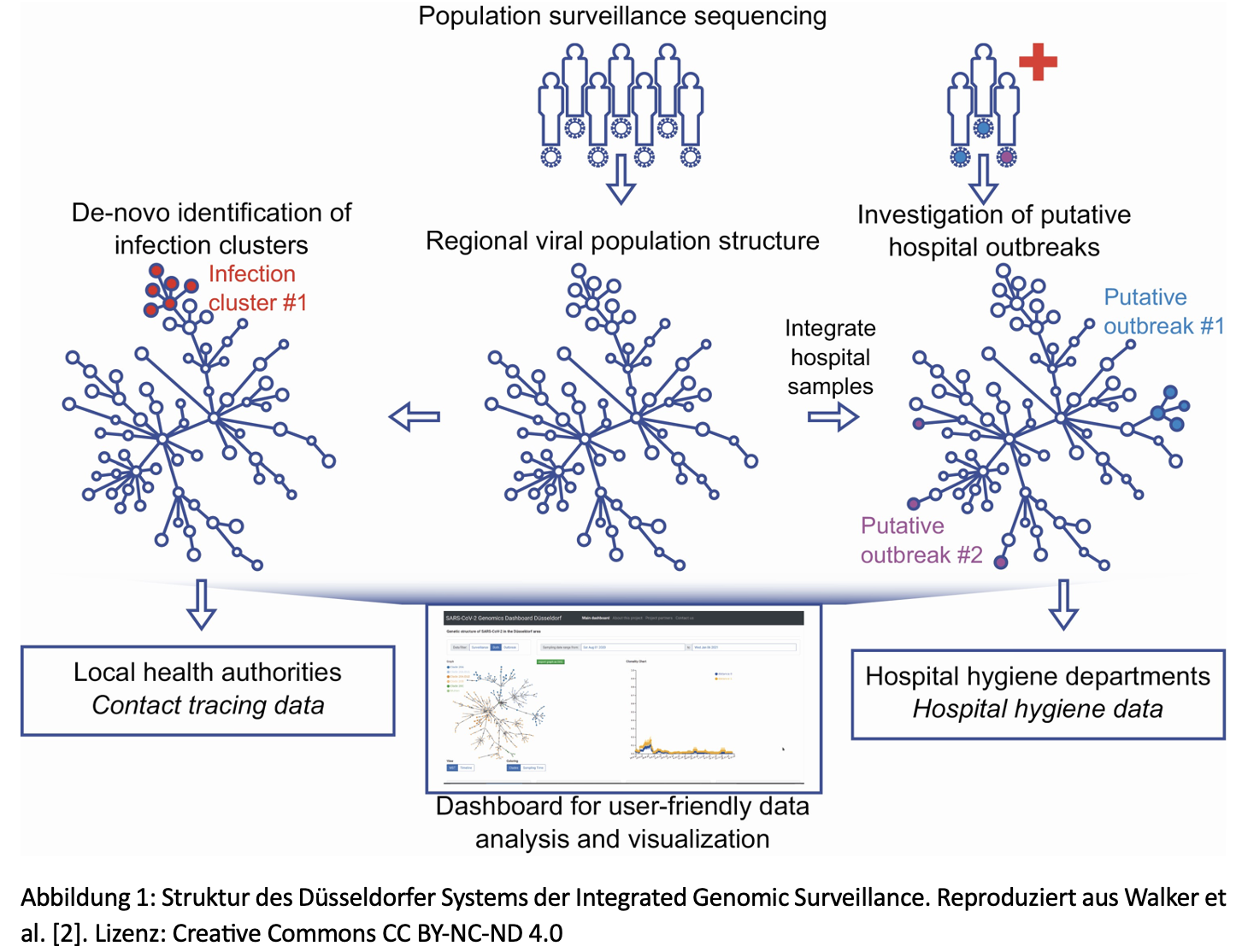
Im ersten Schritt basiert das System darauf, einen so großen Anteil der in Düsseldorf zirkulierenden SARS-CoV-2-Coronaviren wie möglich zu sequenzieren – über 2021 hinweg in der Regel zwischen 40 und 50 % aller PCR-positiven Corona-Tests, was deutlich höher ist als die damals geltende bundesweite Corona-Sequenzierungsquote (5 %). Basierend auf den Ergebnissen dieser Sequenzierung werden im viralen Erbgut vorhandene Mutationen identifiziert. Vereinfacht gesagt bilden die Mutationen eine Art „Barcode“, der einzelne Kopien des SARS-CoV-2-Coronavirus voneinander unterscheidet. Je näher zwei virale Proben im Rahmen einer Infektionskette zusammenhängen, desto ähnlicher sind die „Barcodes“ – bei direkten Übertragungen des SARS-CoV-2-Coronavirus zwischen zwei Personen kommt es z. B. in der Mehrzahl der Fälle zu gar keinen oder höchstens einer Mutation, sodass die „Barcodes“ der in den beiden Personen gefundenen Viren bei direkten Übertragungen in der Regel sehr ähnlich oder identisch sind.
In einem zweiten Schritt werden die Mutationsmuster aller sequenzierten SARS-CoV-2-Coronaviren aus Düsseldorf miteinander verglichen und es wird nach Gruppen – sogenannten „potentiellen Infektionsclustern“ – von Viren gesucht, die eine große paarweise Ähnlichkeit – d. h. ähnliche „Barcodes“ – aufweisen. Wenn dabei die viralen Proben zweier positiv auf SARS-CoV-2 getesteter Personen im selben Cluster auftauchen, ist das ein Anzeichen dafür, dass es zwischen diesen eine direkte Ansteckung oder zumindest einen nahen Infektionskettenzusammenhang – z. B. über eine dritte Person, mit der beide Personen in Kontakt waren – gegeben haben könnte.
Im dritten und letzten Schritt werden die gefundenen „potentiellen Infektionscluster“ durch das Düsseldorfer Gesundheitsamt analysiert und es wird nach potentiellen Ansteckungspunkten zwischen den Personen gesucht, deren virale Proben im selben Cluster auftauchen. Dieser Schritt basiert auf sogenannten „strukturierten Interviews“ ebenso wie auf Daten der Kontaktnachverfolgung, die routinemäßig durch das Gesundheitsamt gesammelt werden.
Durch die Kombination der verschiedenen Datenquellen – genetischer Daten mit klassisch-epidemiologischen Daten – lassen sich Infektionsketten in sonst unerreichter Auflösung und Genauigkeit charakterisieren. Dies betrifft insbesondere Ansteckungen, die allein auf Methoden der klassischen Kontaktnachverfolgung basierend nicht detektierbar wären – z. B., wenn es zu einer Ansteckung zwischen zwei einander unbekannten Personen im öffentlichen Raum oder in der Gastronomie kommt. Weiterhin erlaubt die Integration genetischer Daten eine vertiefte Analyse und Qualitätskontrolle der im Rahmen der Kontaktnachverfolgung gesammelten Daten – so z. B. Aussagen dazu, in wie vielen Fällen ein direkter Kontakt auch tatsächlich zu einer Ansteckung geführt hat.
Bereits im letzten Jahr konnten die Düsseldorfer Forscher*innen zeigen, dass das entwickelte System eine effektive Überwachung von genetischen Varianten von SARS-CoV-2 ermöglicht und das Potential zu einer verbesserten Analyse von Infektionsketten hat [2]. Basierend darauf demonstriert die aktuelle Publikation den Einsatz des Systems für die Infektionskettennachverfolgung und zeigt, dass das System auch die Nachverfolgung komplexer Infektionsgeschehen in der Bevölkerung ermöglicht.
Konkret beschreiben die Forscher*innen die Untersuchung von zwei großen potentiellen Infektionsclustern, die im Sommer 2021 – d. h. zu einer Zeit, als „Delta“ noch die vorherrschende virale Variante von SARS-CoV-2 war – in den Düsseldorfer Sequenzierungsdaten detektiert wurden. Die Untersuchung der Cluster, die über den betrachteten Zeitraum hinweg mit ca. 10 % aller Düsseldorfer Corona-Fälle genetisch in Verbindung gebracht werden konnten, zeigte, dass in beiden Fällen ein Zusammenhang mit von Reiserückkehrer:innen importierten Infektionen mit hoher Wahrscheinlichkeit nachgewiesen werden konnte.
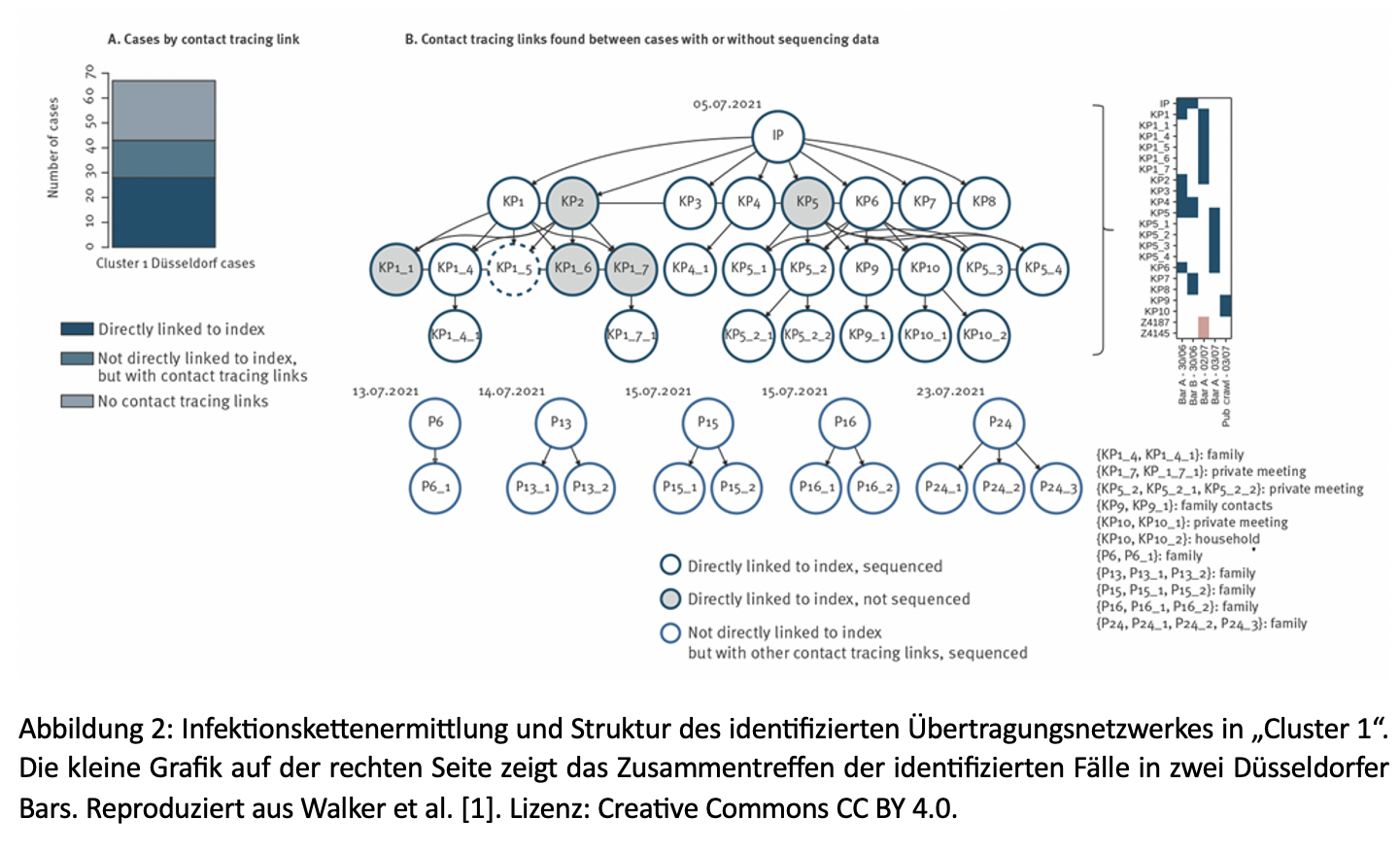
Im Falle von „Cluster 1“, d. h. dem ersten der beiden detektierten potentiellen Infektionscluster, konnte gezeigt werden, dass der Cluster wahrscheinlich auf eine bei Einreise unbekannte importierte Infektion zurückgeht, die sich im Rahmen eines komplexen Übertragungsnetzwerkes u. a. in der Düsseldorfer Altstadt in die lokale Bevölkerung ausbreitete. Konkret gelang es durch das Düsseldorfer System, einen direkten Zusammenhang zwischen einem einzelnen bei Einreise unerkannten SARS-CoV-2-Fall und mindestens 28 SARS-CoV-2-Folgeinfektionen in der Stadt herzustellen (siehe Abbildung 2); diese hätten sich durch z. B. einen PCR-Test bei Einreise höchstwahrscheinlich verhindern lassen. Weiterhin zeigte die Untersuchung des Clusters, dass in mindestens einer der im Übertragungsnetzwerk identifizierten Bars zu der Zeit geltende Regeln zur Corona-Infektionsprävention nicht eingehalten wurden.
Im Falle von „Cluster 2“, d. h. dem zweiten der beiden detektierten potentiellen Infektionscluster, zeigte die Analyse ein diffuseres Infektionsgeschehen, das aber ebenfalls mit von Reiserückkehrern importierten Infektionen und einer möglichen weiteren Ausbreitung in der Düsseldorfer Altstadt in Verbindung gebracht werden konnte. Dabei stellte sich heraus, dass ein signifikanter Anteil der identifizierten Reiserückkehrer*innen vorher in der Region um Barcelona gewesen war; in Zusammenarbeit mit Forscher*innen aus Spanien konnte weiterhin gezeigt werden, dass nah verwandte SARS-CoV-2-Viren tatsächlich während der fraglichen Periode in Barcelona zirkulierten.
Eine wichtige Erkenntnis aus der aktuellen Publikation ist, dass weder genetische Daten noch epidemiologische Daten (z. B. aus der Kontaktnachverfolgung) für sich allein genommen für das Verfolgen von komplexen Infektionsketten ausreichend sind – dies gelingt erst durch die Integration der beiden Datentypen.
Weiterhin illustriert die Veröffentlichung die potentiellen Vorteile einer verbesserten Infektionskettennachverfolgung: das Erkennen von „Hot Spots“, an denen es zu besonders vielen Infektionen kommt, ebenso wie eine Verbesserung der Messbarkeit von z. B. dem Einfluss von Reiserückkehrern oder bestimmten Infektionskontexten wie der Gastronomie. Dadurch könnte perspektivisch eine verbesserte Steuerung von Maßnahmen zur Infektionsprävention erreicht werden – konkret weisen die untersuchten Cluster z. B. auf die möglichen Vorteile von SARS-CoV-2-Tests für Reiserückkehrer oder im Nachtleben hin.
Das Düsseldorfer System war bis Ende 2021 im Vollbetrieb. Die nächsten Schritte im weiterhin laufenden Forschungsprojekt sind vertiefte Analysen der 2021 erhobenen Daten; eine Erweiterung des Systems auf weitere Krankheitserreger; sowie eine Verfügbarmachung der entwickelten Ansätze und Technologien auch für andere Standorte und Städte in Deutschland. Diese Aktivitäten werden gebündelt und koordiniert u. a. über das Netzwerk Universitätsmedizin und darin insbesondere in der GenSurv-Infrastruktur und dem MolTraX-Projekt – mit dem übergreifenden Ziel, zum verbesserten Management der gerade noch ablaufenden und zur Vorbereitung auf mögliche weitere Pandemien beizutragen.
Zitationen:
- Houwaart T, Belhaj S, Tawalbeh E, Nagels D, Fröhlich Y, Finzer P, Ciruela P, Sabrià A, Herrero M, Andrés C, Antón A, Benmoumene A, Asskali D, Haidar H, von Dahlen J, Nicolai J, Stiller M, Blum J, Lange C, Adelmann C, Schroer B, Osmers U, Grice C, Kirfel PP, Jomaa H, Strelow D, Hülse L, Pigulla M, Kreuzer P, Tyshaieva A, Weber J, Wienemann T, Kohns Vasconcelos M, Hoffmann K, Lübke N, Hauka S, Andree M, Scholz CJ, Jazmati N, Göbels K, Zotz R, Pfeffer K, Timm J, Ehlkes L, Walker A, Dilthey AT; German COVID-19 OMICS Initiative (DeCOI); German COVID-19 OMICs Initiative (DeCOI). Integrated genomic surveillance enables tracing of person-to-person SARS-CoV-2 transmission chains during community transmission and reveals extensive onward transmission of travel-imported infections, Germany, June to July 2021. Euro Surveill. 2022 Oct;27(43). doi: 10.2807/1560-7917.ES.2022.27.43.2101089. PMID: 36305336.
https://www.eurosurveillance.org/content/10.2807/1560-7917.ES.2022.27.43.2101089
- Walker A, Houwaart T, Finzer P, Ehlkes L, Tyshaieva A, Damagnez M, Strelow D, Duplessis A, Nicolai J, Wienemann T, Tamayo T, Kohns Vasconcelos M, Hülse L, Hoffmann K, Lübke N, Hauka S, Andree M, Däumer MP, Thielen A, Kolbe-Busch S, Göbels K, Zotz R, Pfeffer K, Timm J, Dilthey AT; German COVID-19 OMICS Initiative (DeCOI). Characterization of Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2) Infection Clusters Based on Integrated Genomic Surveillance, Outbreak Analysis and Contact Tracing in an Urban Setting. Clin Infect Dis. 2022 Mar 23;74(6):1039-1046. doi: 10.1093/cid/ciab588. PMID: 34181711; PMCID: PMC8406867.
https://academic.oup.com/cid/article/74/6/1039/6310838?login=false